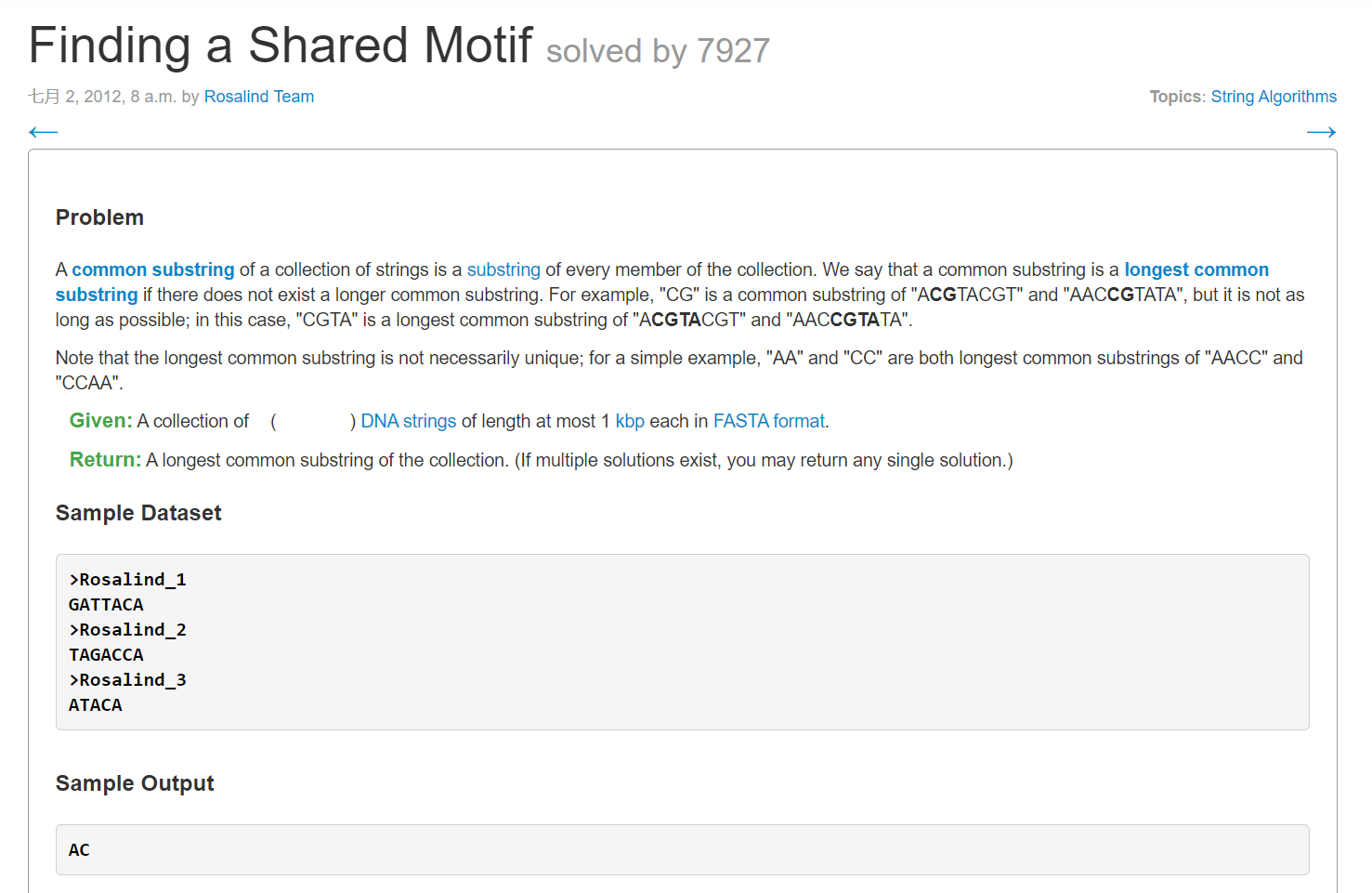
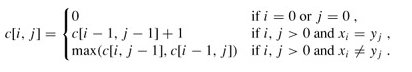多条序列连续公共子串
一、背景
这道题是ROSALIND上的一道题,感觉挺有趣的,就整整。题目放下面了:
在整这道题之前,先来点预备知识,在寻找多序列的公共子串之前,先了解一下两条序列的最长公共子序列,这个是基础知识。在Leetcode上有这样的一道题:1143. 最长公共子序列,可以先解出一下这道题,再来看我们的这个问题。
1.1最长公共子序列
大噶可以先去B站看下别人的解题思路,比如这个小姐姐的,题目如下:
先放上小姐姐的 java 代码:
public int longestCommonSubsequence(String text1, String text2) {
int m = text1.length(), n = text2.length();
int[][] dp = new int[m + 1][n + 1];
for (int i = 0; i < m; i++){
for (int j = 0; j< n; j++){
char c1 = text1.charAt(i), c2 = text2.charAt(j);
if(c1 == c2){
dp[i + 1][j + 1] = dp[i][j] + 1;
} else {
dp[i + 1][j + 1] = Math.max(dp[i + 1][j], dp[i][j+1]);
}
}
}
return dp[m][n];
}在放上对应的 python3 代码:
def longestCommonSubsequence(self, text1: str, text2: str) -> int:
m, n = len(text1), len(text2)
dp = [[0]*(n+1) for i in range(m+1)]
for i in range(m):
for j in range(n):
if text1[i] == text2[j]:
dp[i+1][j+1] = dp[i][j] + 1
else:
dp[i+1][j+1] = max(dp[i+1][j], dp[i][j+1])
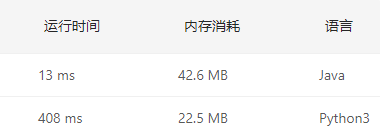
return dp[m][n]再看一下两种语言的速度差距(所以该学什么语言不用我多说了吧!+ 。+):
1.2最长公共子串
子串和子序列是有差别的,子串指的是字符串中连续的一段字符,而子序列的话是没有这个要求的,其实这两种的解题思路是一样的。子串的数学表达式为: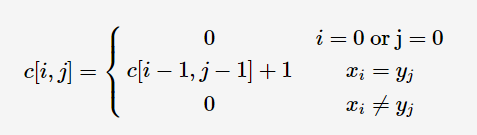
在有前面的基础上,我们直接放代码:
def Find_Lcsubstr(s1, s2):
m, n = len(text1), len(text2)
dp = [[0 for i in range(n+1)] for j in range(m+1)]
max1 = 0
state = 0
for i in range(m):
for j in range(n):
if text1[i] == text2[j]:
dp[i+1][j+1] = dp[i][j] + 1
if dp[i+1][j+1] > max1:
max1 = dp[i+1][j+1]
state = i + 1
return s1[state-max1:state], max1 下面我们尝试寻找两条DNA序列的最长公共子串:
data = """>Rosalind_2556
ATGTGAGGGATGTTAGCTTAGTCCGGAGATGCGATGCGGATAGCGTCCAAAGGACATCGT
GGGAAGTAACATCTGTGCTGAGTGAGTTCAACATGCAGGAAGCCCTGGTTATAGCCTACA
AACGCACGCCTTGTATCTACCGCTCACGGCGTTAGGAGTTTATGACCATGACTGGTCCTC
TGGACCTGGCCTGCGTGTGGGGTTCCGTCCGAGCAGAGGATCTTCATCTAGTCCTAATCG
CCGACTGCTTAGTCTGATGTTATCGAATGACTTAAACCGCTTTGGGGTTAGGCGGCGTCT
ACTCATGAGGTACTTACCTTGCTAGGCATGACGTTCTTGATTTAGGCACAGTCGCATTAG
TGCCATGGGTTGGGACTAAGATCACTTTCCGTCACATGATAGCACACCTGAACTTTCTAG
TCGTAAACCTACTAGAATGGATCACGCCACTCGGCCTCGGGTAAGATCCCAATGCCACTT
CCCACTCCTACGAAGCAAGGGTGTACACTCTTTTGCCCTGACATCCGCCCTCTTGCGAAC
CGCCCGGGGATACTCGCCCCGCCTTCGAGTGGCATGAAGCCAGTTAGATTTACCTGGGTA
TAAGTGTGTTCTTCGGGGAGCGAGCAGCTTATGAAGGGTCAGGTTGTTCAAAAACCAGGC
AGTGGGAGCCACAGGCCACCTGTGCGCTAGAAAAATACCGTGTGCAACAATCTCTATGCG
AGACCACTTCTAATTAAAATCGCAAAAGATAACCGTCCGGGATCGGCTGCTGCCTGCGGC
GATGTGTGTGCCCCCCATACGATGCTTTCGTATTACATGCTTAAGGAATGACAAGGTGCC
CACTGGGTGAAACATTCGTAGTCTCAGCATTTGCGTCGGCTGTTTCTCACTAGGCTGCCA
GAAACTGGTACGTGGACAAACTATCCTTTTTTGAGAGGGACACGACTTTACGCCCGGACC
CTGCACAGCCGCATGTATCTCCTCCCTTGGGCTATTAGAC
>Rosalind_8024
ACACATACTCGCTGGCAATGGCTTCTGCTGACACAGATCGCGTTTATTGCCCTGACATCC
GCCCTCTTGCGAACCGCCCGGGGATACTCGCCCCGCCTTCGAGTGGCATGAAGCCAGTTA
GATTTACCTGGGTATAAGTGTGTTCTTCGGGGAGCGAGCAGCTTATGAAGGGTCAGGTTG
TTACAGCGCTCACGTTATATATGTAACGGAATGAACCTTCGTGCGTTCTCACCTCAGATT
ATAAAGTCCCGTGAACCGGACAATGTGGCCAAAAGTCCTGGGCGTTGCAATGATAGCTCC
TCGGCAGTATGCTGAAGGGGAAAAGCTTACCTGTTCTATAGTATCGTACTCAACCGCCGG
CCTCAGCGCTTCGTCCAGGGTGCGCCGGGAATCAGTGTCTAAGGGCTCAACTCGCTCATG
GAGGTTACGACAACGACTCTGAGGCAATCCCATCGGTTAATGCACAACACCGAGGCGGGG
GGGCATGAGTTTGACGATACCCATGTAAGAACAACGTCCGATTGCCCAAGCACCTAGTGG
GACTTGGTCTCGCTCCTCCGATAAATAGTACGGCCACTCAATCTTTGGGATCCAGGTCAA
CGCCTGATCACGGTCCCAGCTGAACGTAGGAGATGGTCGACACTTACGGCTTAATATTAC
GTTACTTACTATCGGTTGCTAACGTGGACCCCCTATCCCTGAGCCTTTTGGCTCAGTTAA
AATAATCCTTGTTACGGTGGGTAACGGCGGTTCATACAACTTCGAGCTCTCGATTCGAGT
AGGGCCTGATCTAAAGTGAAATCGACTCGGCATGGGTTGAAACTACCTATAATCAAGTGG
CGAATGAAGAGGCAGTTCACATCTGGTGATATTTCAGTTTCATGGGTTCTTAGACGAGGG
CGTTCTTATCTTCGCACAGCGCTCCTCCTAGATCATACGTTAGCCCTGGTCTCAGAAACA
TGGCAAGAAGGCTCCCTCGGACGAGGCGGTCTAATTAAGT
"""
def Find_Lcsubstr(s1, s2):
m, n = len(s1), len(s2)
dp = [[0 for i in range(n+1)] for j in range(m+1)]
max1 = 0
state = 0
for i in range(m):
for j in range(n):
if s1[i] == s2[j]:
dp[i+1][j+1] = dp[i][j] + 1
if dp[i+1][j+1] > max1:
max1 = dp[i+1][j+1]
state = i + 1
return s1[state-max1:state], max1
def main():
seq_lines = data.split('>')[1:]
seq = []
for i in seq_lines:
res = i.split('\n')
string_line = ''.join([j for j in res[1:]])
seq.append(string_line)
print(Find_Lcsubstr(seq[0], seq[1]))
main()
# 输出为'TTGCCCTGACATCCGCCCTCTTGCGAACCGCCCGGGGATACTCGCCCCGCCTTCGAGTGGCATGAAGCCAGTTAGATTTACCTGGGTATAAGTGTGTTCTTCGGGGAGCGAGCAGCTTATGAAGGGTCAGGTTGTT', 136二、多条序列的连续公共子串
好了,我们已经会两条序列的寻找呢,在进行多条序列的公共子串分析之前,我们应该有这样的一个概念:两条序列的公共最长子串直接一比较就行了,那么如果是多条呢?是不是需要多条序列之间的两两比较呢?那这个该有多复杂呢?
其实有个简单的想法,那就是先随便的挑出两条出来,把这两条比较一下,因为不知道最后结果序列会有多长,可能你在这两条序列中找到的最长的子串,可能在别的序列中就没有那么长了,所以应该在挑出来比较的两条序列中把一定长度的所有公共子串都拿出来,最后想要的多条序列的最长公共子串一定在前面两条公共子串的子集中,可能是子集,也可能是某一个子集的子串(不知道这里搞明白没有)。
举个简单的例子:假设啊,假设,你在挑出的两条序列的比较中得到了符合一定长度的两个公共子串:[‘TTGCCCTGACATCCGCCCTCTTGCGAACCGCCCGGGGATACTCGCCCCGCCTT’, ‘CTTCGGGGAGCGAGCAGCTTATGAAGGGTCAGGTTGTT’],然后你用这两条序列去分别的和除这两条序列以外的所有序列比较,运气好可能都是其他DNA序列的子串,这个时候我们就可以选择最长的那条,运气不好的情况下是什么样的呢?假设str1 = ‘TTGCCCTGACATCCGCCCTCTTGCGAACCGCCCGGGGATACTCGCCCCGCCTT’这条子串在和第三条DNA序列比较的时候只有前面20个字符是公共且最长的,那么我们在这个时候就应该把原来的全长子串,变成它的前二十个字符,str1 = str1[:20],然后再用这个改变后的str1去和第四条DNA序列去比,如果后面所得长度又发生变化了,再改变str1的值,一直这样迭代下去,这样我们得到的就是所有DNA序列的公共子串了,然后我们把前面两条序列和其他DNA序列都比较一下,会得到一些子串,选出最长的就是我们想要的结果了。
不多BB,我们直接整这道题:
# N个字符串的所有公共子串长度大于阈值的子串
# 设置阈值
MIN_NUM = 8
def get_maxstr(str1, str2):
total_str = []
lstr1 = len(str1)
lstr2 = len(str2)
record = [[0 for i in range(lstr2+1)] for j in range(lstr1+1)]
# 构建打分矩阵
for i in range(lstr1):
for j in range(lstr2):
if str1[i] == str2[j]:
record[i+1][j+1] = record[i][j]+1
# 遍历打分矩阵,获取相应的子串
for i in range(lstr1+1):
for j in range(lstr2+1):
if record[i][j] >= MIN_NUM and +
((i == lstr1 or j == lstr2) or (record[i+1][j+1] == 0)):
len1 = record[i][j]
total_str.append(str1[i-len1:i])
return total_str
def main():
f1 = open('13.input.txt', 'r').read()
seq_lines = f1.split('>')[1:]
seq = []
for i in seq_lines:
res = i.split('\n')
string_line = ''.join([i for i in res[1:] if i])
seq.append(string_line)
# 获取开始两条DNA序列的公共子串 len > MIN_NUM
res_lst = getmaxstr(seq[0], seq[1])
finall_lst = []
# 将前面获取的子串分别与其他的DNA序列比对,获取最长子串
for i in res_lst:
state = True
for j in range(2, len(seq_lines)):
one_ouf = getmaxstr(i, seq_lines[j])
if not one_ouf:
state = False
break
# 得到最长的子串
Long_return = sorted(one_ouf, key=lambda x:len(x))[-1]
if len(Long_return) < len(i):
i = Long_return
if state:
finall_lst.append(i)
print(sorted(finall_lst, key=lambda x:len(x))[-1])
if __name__ == '__main__':
main()
这段代码是年前写的,现在看的话有非常大的提升空间,感兴趣的话可以自己尝试下怎么加快速度!!