真菌线粒体基因组的组装及注释
最近在做真菌基因组的组装注释工作,发现之前组装的线粒体基因组有点问题(基因注释只注释出来 rRNA,长度也不太对),然后重新组装注释一下。主要还是利用NCBI上已有的Alternaria mitochondrion做为参考序列进行组装。
0.环境的配置
conda create -n mitogenome sra-tools fastp -y -c bioconda
conad activate mitogenome
# MITGARD 用来有参拼接线粒体基因组
# MITGARD: https://github.com/pedronachtigall/MITGARD
cd /data/chaofan/software/
git clone https://github.com/pedronachtigall/MITGARD.git
cd MITGARD/bin
# 加入PATH 运行过程中需要依赖到一些脚本文件,不放入PATH会报错
export PATH="/data/chaofan/software/MITGARD/bin:$PATH"
# MITGARD有好几个软件依赖 不装用不了
# 依赖的软件自行配置,都能通过conda安装特别注意, MITGARD.py、msa2consensus.py、sam2msa.py、RearrangementCheck.py 这几个脚本的解释器地址要改成当前环境的python解释器,不然会报错。 把脚本文件的第一行从/usr/bin/python 改成/usr/bin/env python。
1.数据的下载及预处理
# prefetch 和 fastq-dump都属于sra-tools工具包,直接conda安装就好了
prefetch SRR12578435 -O ./
# 解压sra文件为fastq
fastq-dump --split-3 SRR12578435/SRR12578435.sra
# fastp 去除接头及低质量碱基
fastp -i SRR12578435_1.fastq -o SRR12578435_f1.fastq -I SRR12578435_2.fastq -O SRR12578435_r2.fastq --thread=4 --length_required=40 -j SRR12578435.json
# ncbi上线粒体基因组就自己手动下载了2.MITGARD 组装线粒体基因组
软件运行很简单,前面的注意事项弄完一般就不太会报错了。
/data/chaofan/software/MITGARD/bin/MITGARD.py -s Z7 -1 SRR12578435_f1.fastq -2 SRR12578435_r2.fastq -R PN2.faZ7_mitogenome.fa就是我们的结果文件了,简单的less查看一下,看看是否有大量的N,有大量N就不行了,可能是你选的参考线粒体基因组不对。
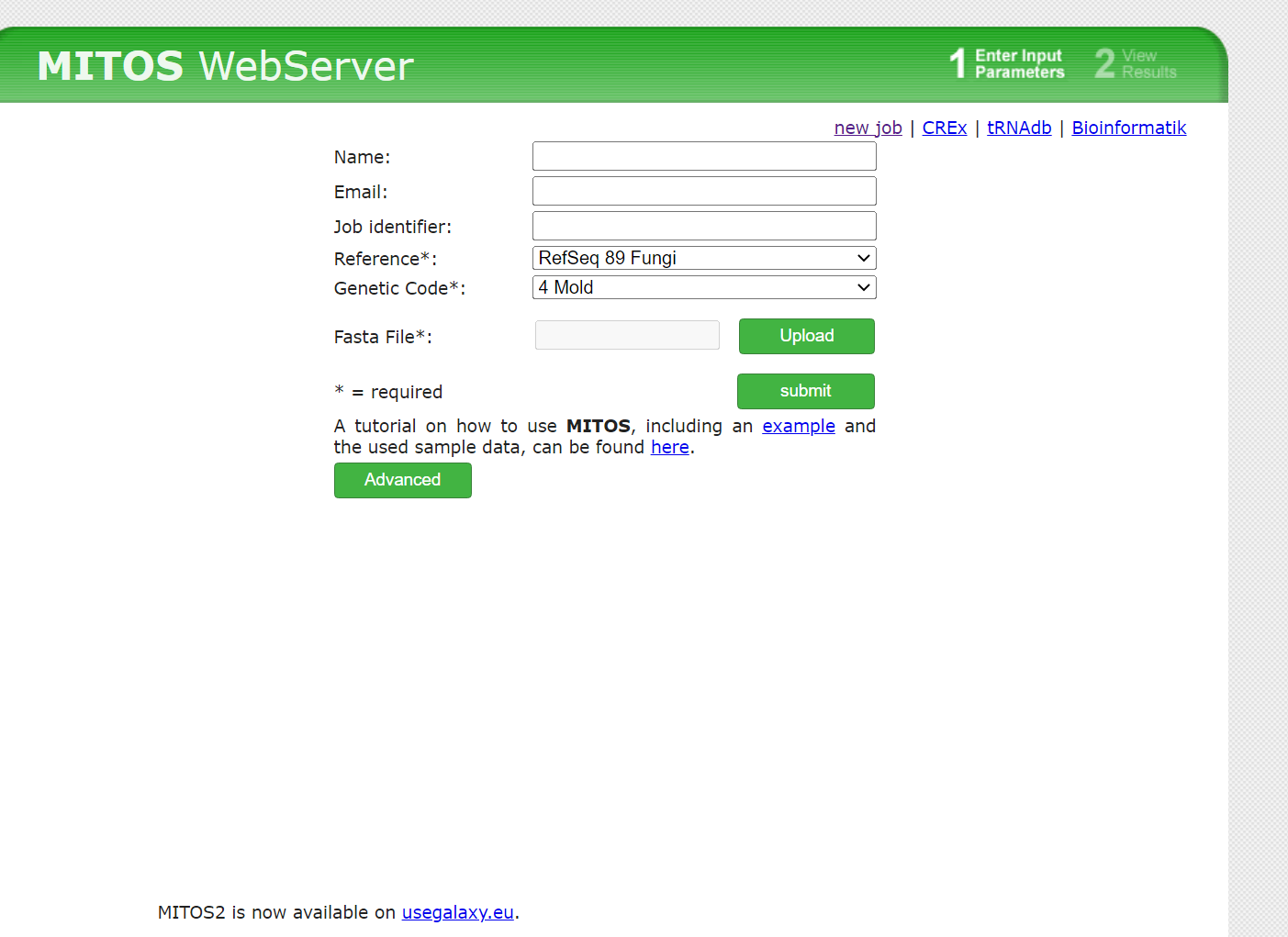
3.MITOS在线注释线粒体基因组
MITOS的使用非常简单,这里选择真菌,密码子表选择4就好了。速度也非常快,毕竟线粒体基因组才50Kbp长,基因也非常保守。