0.前言
在做真菌的一些生信分析中,可能需要真菌的繁殖方式的信息。真菌的繁殖方式主要有两种:同宗配合与异宗配合,自然界中绝大多数的情况下都是异宗配合。控制真菌交配型的基因主要有两种: MAT1-1 和 MAT1-2,异宗配合指的是繁殖时必须需要这两种不同的交配型,同宗配合则没有这个限制1。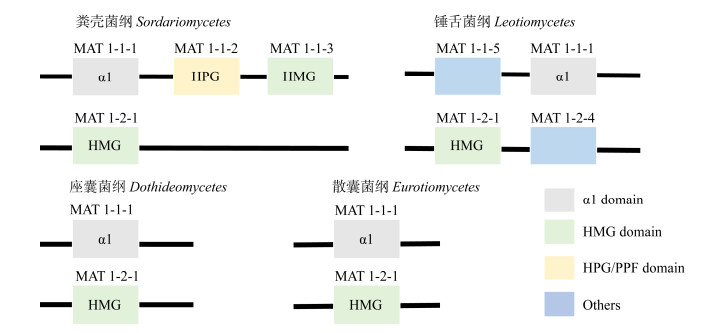
1.NCBI 下载同源序列
通过上面的知识,我们了解到Alternaria属于Dothideomycetes,主要有两种交配型基因MAT 1-1-1与MAT 1-2-1。然后我们使用 NCBI 进行检索,关键字为((Alternaria) AND mating-type) NOT partial,发现搜索结果也主要的分为了MAT 1-1-1与MAT 1-2-1这两种。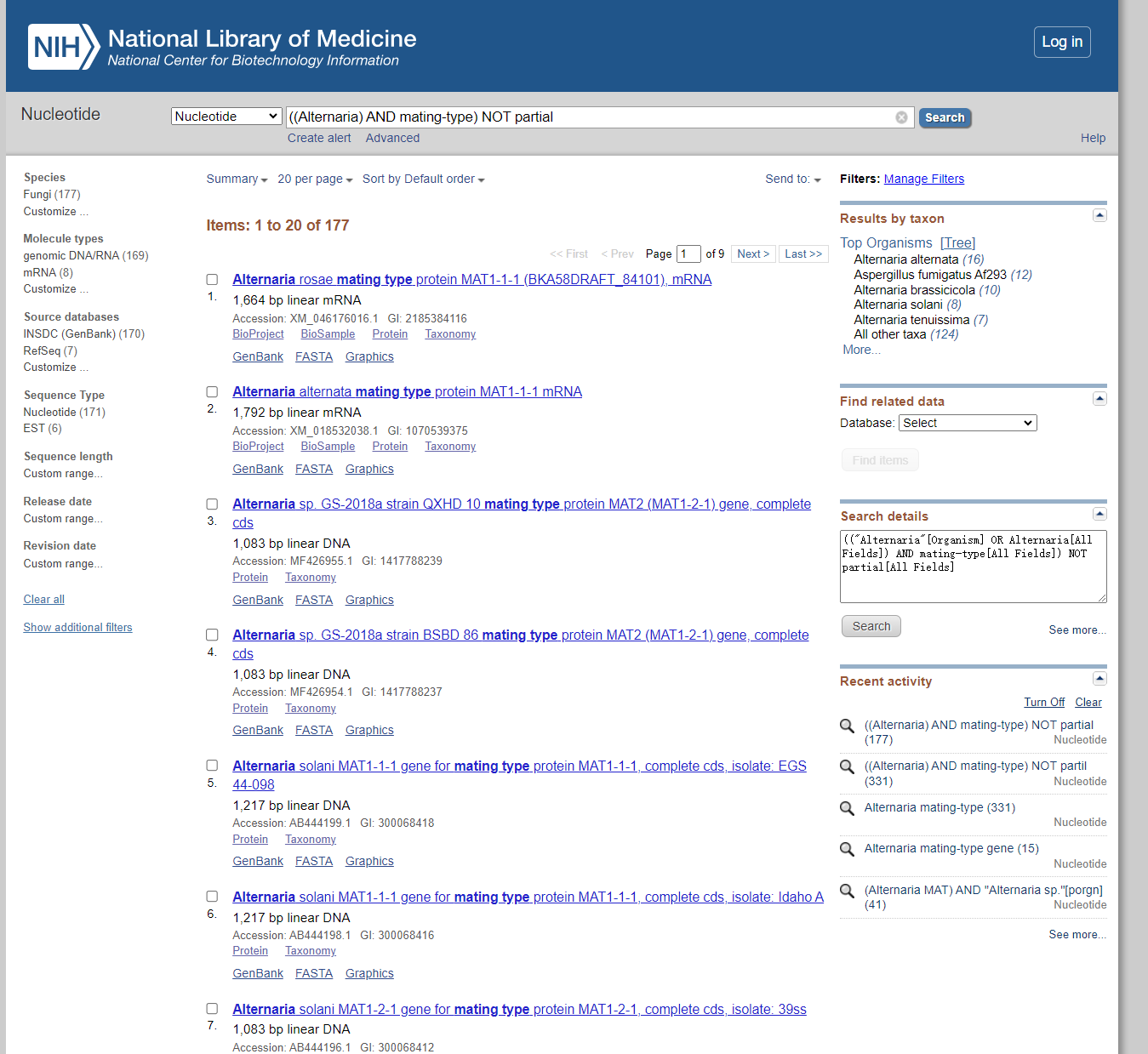
我们两种交配型分别挑选 6~7 条 items,然后按顺序点击下图标签,这里第四步可以选择下载 DNA 还是 protein 序列,我们直接选择 DNA 序列。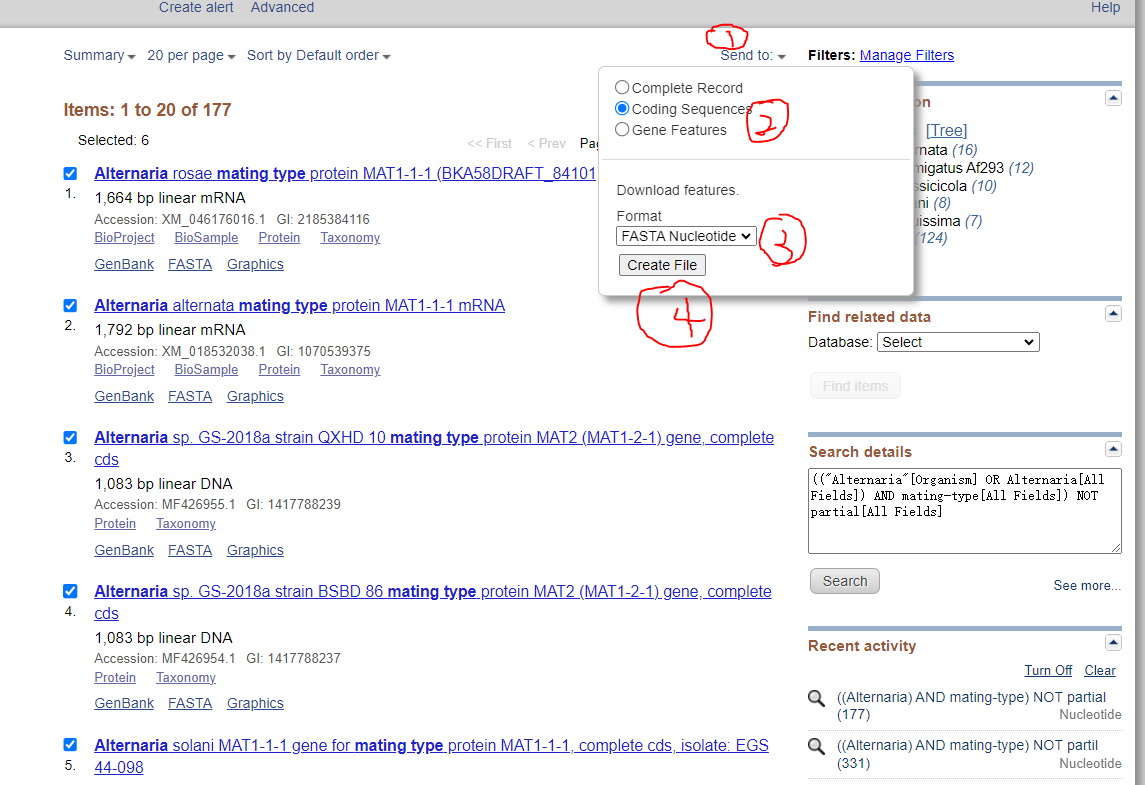
交配型 MAT1-1 与 MAT1-2 的序列差异非常的大,不管是 DNA 还是 protein,你也可以自己试试。
2.前处理
主要使用 blastn 来鉴定,输入数据包括上一步下载的 DNA 序列和组装好的基因组,如果你的物种基因组很小,就几十 Mb,然后你的二代数据用SPADEs能组装的非常好。用SPADEs怎么组装基因组之前写过,这里就不详细讲了。
上一步下载的 fasta 格式的 DNA 序列的 ID 信息有点乱:
>lcl|AB444193.1_cds_BAJ10530.1_1 [gene=MAT1-1-1] [protein=mating type protein MAT1-1-1] [protein_id=BAJ10530.1] [location=join(<1..142,190..>631)] [gbkey=CDS]
最好修改下 ID,方便后续处理,我的话会改成:
>ADE44136.1__MAT1-1-1
>ADE44135.1__MAT1-1-1
>ADE44134.1__MAT1-1-1
>ADE44132.1__MAT1-1-1
>ADE44131.1__MAT1-1-1
>ADE44128.1__MAT1-1-1
>ADE44126.1__MAT1-2-1
>ADE44125.1__MAT1-2-1
>ADE44124.1__MAT1-2-1
>ADE44123.1__MAT1-2-1
>ADE44120.1__MAT1-2-1
>ADE44118.1__MAT1-2-13.交配型的鉴定
3.1 blast 建库
怎么安装 blast 什么的这里也不讲了,自己百度。
makeblastdb -dbtype nucl -in Alternaria_sp.nr.re.cds -out Alternaria_sp.nr.re.cds3.2 blastn 比对
blastn -query sample1.geno.fa -db Alternaria_sp.nr.re.cds -outfmt 6 -evalue 1e-10 -max_target_seqs 12 -num_threads 10 -out sample1.MAT.blasn结果就两行:
NODE_11_length_1128118_cov_18.265760 ADE44132.1MAT1-1-1 99.784 925 2 0 698132 699056 1170 246 0.0 1698
NODE_11_length_1128118_cov_18.265760 ADE44132.1MAT1-1-1 99.592 245 1 0 699104 699348 241.99e-125 448
因为我们使用的是 mRNA 剪切过后的 CDS 序列,所以比上的区域有个大的 GAP。这里最好将-max_target_seqs设为你之前从 NCBI 上下载的总序列数,有可能会鉴定到两种交配型的情况,这种很罕见,但有,处理的时候注意。
References:
https://wswxtb.ijournals.cn/html/wswxtbcn/2020/5/tb20051572.htm

